Primero que todo debo contarles que ya esta lista la nueva version de R (2.12.1) y es descargable desde este link, como siempre free o al gratin, como nos gusta la cerveza. http://cran.cnr.berkeley.edu/
Esta ultima version esta disponible para todos los sistemas operativos y no tiene mas de un mes de haber visto la luz.....
Por otro lado estuve pensando (porque aveces tengo ideas) y creo que lo mejor es empezar por una prueba bien basal, superbasal, ya que intente explicarle a un amigo algo basico de R y aun cree que es mas facil la calculadora de hacer cuentas del fiado de la tienda!!!. Pero de todos modos siento ahora que tuve la culpa porque trate de explicarle que es "linea de comandos" con una simple suma con los argumentos:
> a<- 4+4
> a
[1] 8
Simplemente el tomo la calculadora y le dio 4+4= 8.
Asi que ahora creo que lo que queria explicarle necesitaba hacerlo de otra manera; por ende la primer prueba que intentare precisar es la prueba de T para muestras independientes, ya que es una prueba ampliamente utilizada en muchos campos (talvez a mi amigo le sirva en su calidad de ingeniero de alimentos, aun mas de lo que le "sirven" los landmarks).
Para empezar dire que es una prueba basica que no tiene muchos requerimientos, solo necesita que los datos tengan varianzas homogeneas y que sean normales, de lo contrario la prueba que se debe aplicar no es la de T sino la prueba de U. Aunque muchas personas afirman que la prueba de T es altamente robusta y puede soportar que las varianzas no sean homogeneas y que no cumplan normalidad. En cualquier caso R provee argumentos para realizar prueba de T para varianzas no homogeneas, por lo cual no hay mucha necesidad de discutir los parametros de los datos para realizar esta prueba.
Entonces lo primero que uno debe hacer es crear su matriz de datos (uno puede crear los datos directamente en R, pero el problema es que es mucho mas facil mantenerlos como tablas .txt o en .csv como matrices cuando uno tiene gran cantidad de datos). Esto se puede hacer con excel o mas facil en documentos de texto y se salvan con el formato deseado. Para este caso utilizaremos el formato .csv que es un archivo separado por comas y hay que aclarar que R utiliza el punto (.) como separacion de decimales y no la coma, ya que con esta el caracteriza las columnas.
entonces en un documento de texto basicamente se escriben los datos:
Pfert,Nfert
48.2,52.3
54.6,57.4
58.3,55.6
47.8,53.2
51.4,61.3
52.0,58.0
55.2,59.8
49.1,54.8
49.9,
52.6,
podemos darnos cuenta que existen dos columnas, estos datos son de unas plantas que han sido tratadas con dos tipos de fertilizantes (P y N) y lo que queremos saber es si existe diferencia entre el largo del tallo de las plantas tratadas con diferente fertilizante.
por esta razon el encabezado de las columnas es Pfert y Nfert. y los datos que se escriben hacia abajo de ellas son la longitud de los tallos de varios individuos tratados.
Al escribir esto en un documento de texto utilizando un blog de notas, geany o cualquier editor de texto simplemente se salva el archivo con el siguiente nombre
ej21.csv
indicando que el archivo esta delimitado por comas.
Una vez creado mi archivo lo pongo en un directorio determinado (podria ser hasta el escritorio) y cambio el directorio de archivos en r.
Esto se hace en windows con el mouse utilizando la barra de herramientas y dandole a cambiar el directorio. y en linux se utiliza el comando:
> setwd (aca va la direccion en donde estan mis datos sobre la cual voy a trabajar en R)
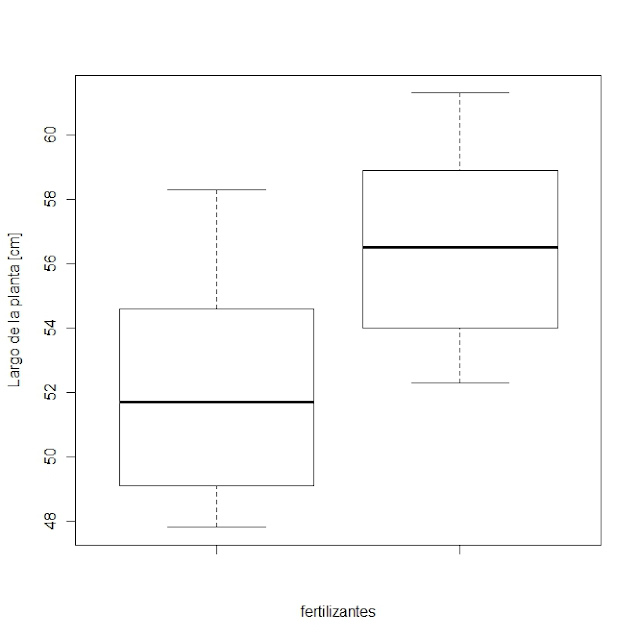
> ej21 <- read.csv("ej21.csv",header=T)#(llamo a R el archivo que cree en formato .CSV (separado por comas) !!se puede crear en excel y guardarlo en formato .csv pero se deben cambiar los ";" por "," y los decimales deben estar separados por "." no por ","en cualquier directorio). >objects()#(asi puedo ver los objetos que he creado en R, que hasta este momento solo seria uno "ej21") >ej21#(asi puedo ver lo que tiene mi archivo ej21 y garantizar que no hayan errores en el archivo) >attach (ej21) #(para tomar mis colomnas como objetos) >boxplot(Pfert,Nfert,ylab="Largo de la planta [cm]", xlab="fertilizantes")#(inspecciono mis datos mediante un grafico apropiado de caja y bigotes) >shapiro.test(Pfert) # (realizo prueba de normalidad para mis datos con el fertilizante P)mis resultados serian:
Shapiro-Wilk normality test
data: Pfert
W = 0.9512, p-value = 0.6833 >shapiro.test(Nfert) #(realizo prueba de normalidad para mis datos con el fertilizante N) mis resultados serian:
Shapiro-Wilk normality test
data: Nfert
W = 0.9705, p-value = 0.9021
>var.test(Pfert,Nfert) #(realizo una prueba de homogeneidad de varianzas, tambien llamada prueba de F para saber si mis varianzas son homogeneas)resultados:
F test to compare two variances
data: Pfert and Nfert
F = 1.149, num df = 9, denom df = 7, p-value = 0.8744
alternative hypothesis: true ratio of variances is not equal to 1
95 percent confidence interval:
0.2382247 4.8224457
sample estimates:
ratio of variances
1.149009 > t.test(Pfert,Nfert,alternative="two.sided",var.equal=T) # (por ultimo realizo mi prueba de T para varianzas homogeneas (si mi prueba de homogeneidad de varianzas me lo indica)(el argumento "var.equal=T" o "var.equal=TRUE" indica que mis varianzas son homogeneas, si no lo fueran se pondria "var.equal=F" o "var.equal=FALSE" )# (y el argumento (alternative="two.sided") se pone cuando quiero realizar una prueba de dos colas, como utilizaremos en este caso.)resultados:
Two Sample t-test
data: Pfert and Nfert
t = -2.9884, df = 16, p-value = 0.008686
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-7.931470 -1.348530
sample estimates:
mean of x mean of y
51.91 56.55
Y listo!!!nos pillamos!!!

Hey! Gracias! La verdad es que apenas soy un novato con R y esto me ha ayudado muchísimo, espero que poronto pongas algo sobre cuartiles y comparaciones múltiples :D
ResponderEliminarMagnífico blog :)